|
全基因组甲基化测序技术介绍
***************************************************************************************************** 技术简介: 全基因组重亚硫酸盐测序(whole genome bisulfite Sequencing)是基于重亚硫酸盐的甲基化分析方法,首先通过重亚硫酸盐对样本DNA进行处理,将未甲基化的C碱基转化为U碱基,而甲基化的C碱基则不会改变,进行PCR扩增后U碱基会变成T,与原本甲基化的C碱基区分开,再结合高通量测序技术,可绘制单碱基分辨率的全基因组DNA甲基化图谱。 应用领域:
技术参数与实验流程
***************************************************************************************************** 技术参数
实验流程 A. 建库测序流程 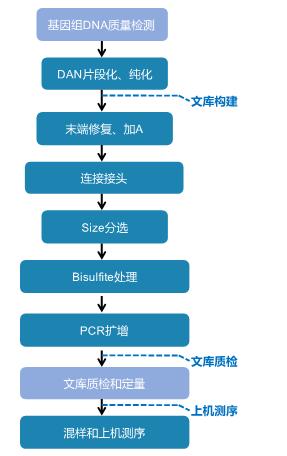 B. 数据分析流程 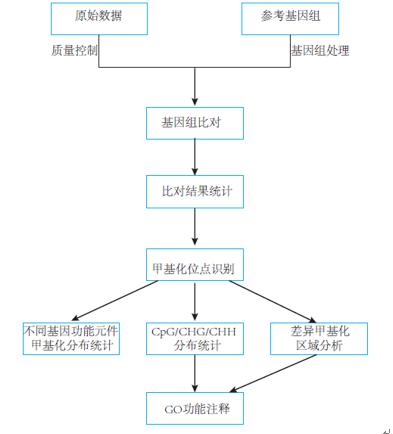
经典文章解读 背景:启动子区DNA甲基化参与转录调控,但甲基化的程度和表达抑制程度的关系仍不明确;此外,启动子区和基因主体区的甲基化功能相关性也不明确。 方法:Trio家系三个样本外周血单核白细胞提取RNA和DNA,分别进行转录组测序和全基因组甲基化测序。 结果:
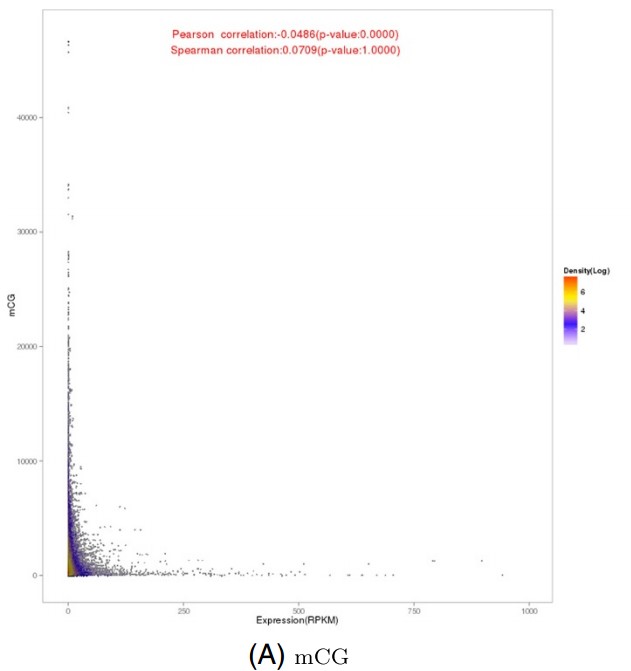
图一:基因表达和甲基化程度呈现明显的L形状模式。即极端高表达的基因甲基化程度很低,极端低表达的基因甲基化程度很高。
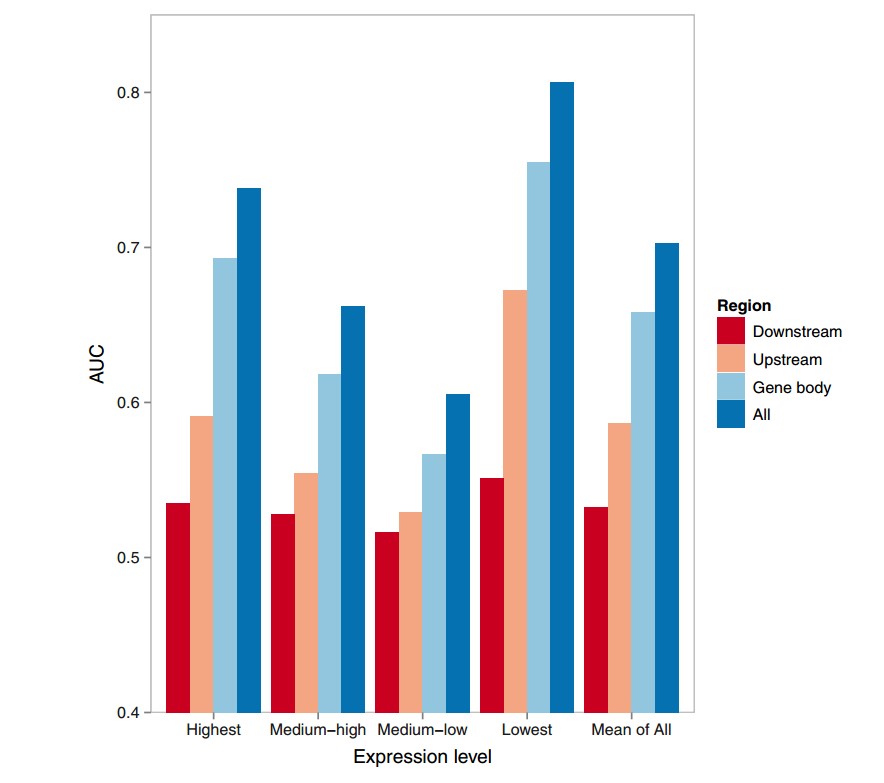
图二:基于随机森林的表达模型发现,对于基因整体表达高低,基因本体甲基化程度比启动子区甲基化程度的指标作用更佳
案例2:番茄果实发育的单碱基分辨率甲基化谱揭示与成熟相关的表观修饰 背景:番茄果实的成熟是由植物激素乙烯引发,但它的影响受一个未知的发育线索所限制,为了确定这个未知的发育线索是否涉及表观遗传重塑,本研究对番茄用甲基转移酶抑制剂处理,结果发现果实提前成熟。通过对对果实发育中从不成熟到完全成熟的4个阶段番茄果实进行全基因组甲基化测序,数据表明,表观基因组在果实发育过程中不能是静态的,可能已被选择用于确保发育过程的保真度。 方法: 研究人员对果实发育中从不成熟到完全成熟的4个阶段番茄果实(17d,39d,42d,52d),2个成熟缺陷变异株 (Cnr和rin变异) 的成熟期果实和1个野生型番茄叶片进行全基因组甲基化测序,测序深度为10×。 结果:
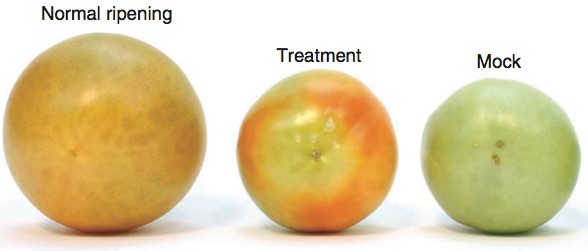 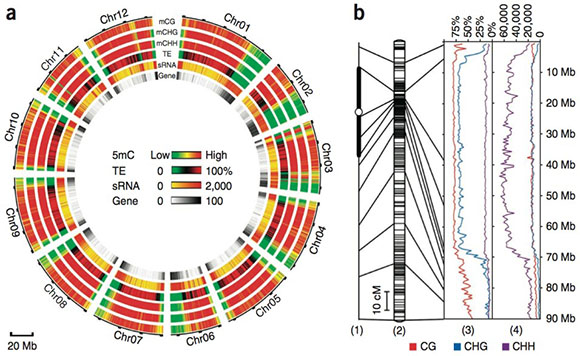
图一: 番茄的表观基因组图谱

图二:不同组织、发育时期的甲基化差异
 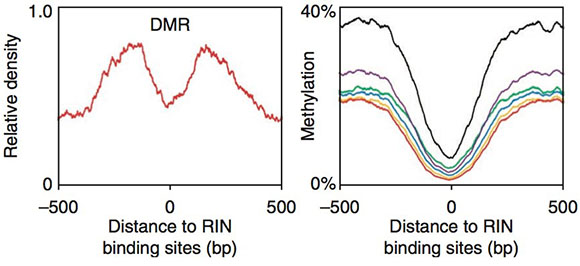
图三: RIN结合位点区域特征
参考文献: Zhang S., et al., Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening. Nature biotechnology, 2013 Feb;31(2):154-9. |








