|
ChIP-seq技术介绍
******************************************************************************************************* 技术简介: 染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的有力方法,通常用于转录因子结合位点或组蛋白特异修饰位点的研究。将ChIP与第二代测序技术相结合的ChIP-Seq技术,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的DNA区段。 ChIP-seq的原理是首先通过染色质免疫共沉淀技术(ChIP)特异性地富集目的蛋白结合的DNA片段,并对其进行纯化与文库构建;然后对富集得到的DNA片段进行高通量测序。研究人员通过将获得的数百万条序列标签精确定位到基因组上,从而获得全基因组范围内与组蛋白、转录因子等互作的DNA区段信息。 应用领域:
技术优势:
技术参数与实验流程
********************************************************************************************************************************* 技术参数
实验流程 A. ChIP-seq建库测序流程 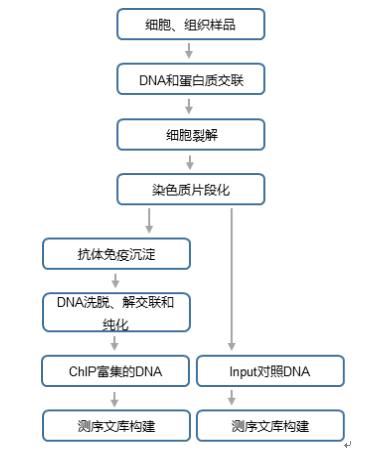 B. 数据分析流程 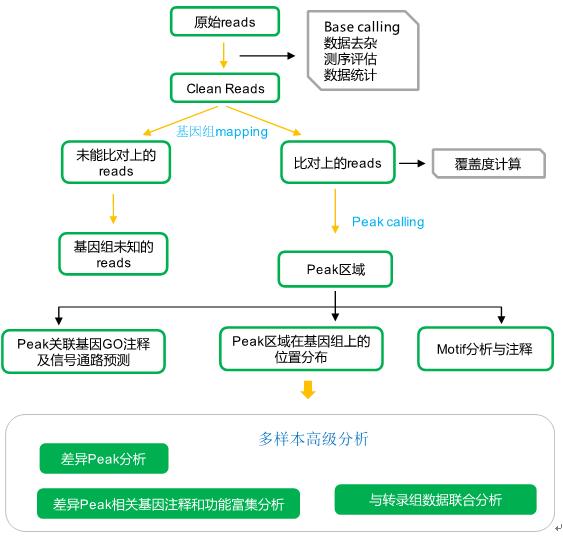
经典文章解读 背景:
方法:
结果:
参考文献
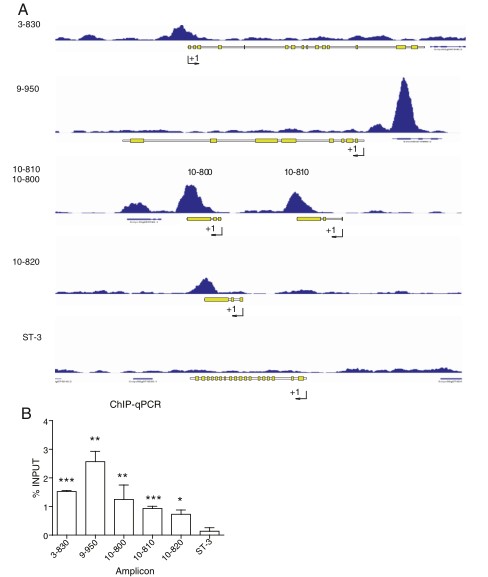
图一:通过ASR1抗体富集到的peak区域,并用qPCR验证转录因子识别位点是否准确
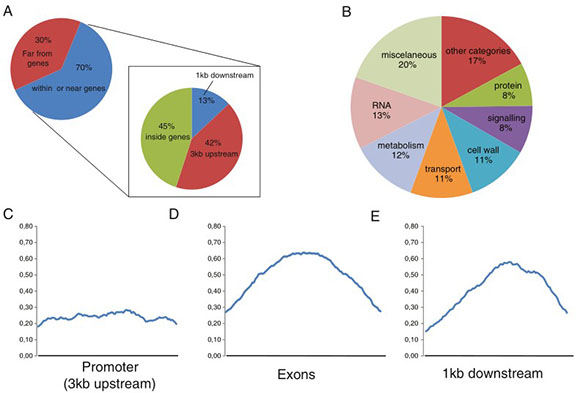
图二:ASR1结合的基因组区域分析
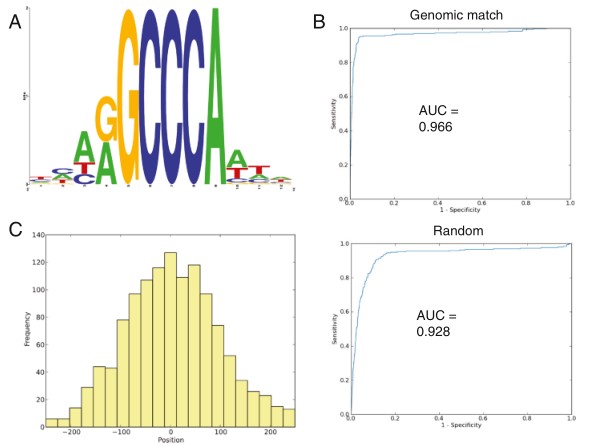
图三:确定了一个ASR1-binding DNA motif
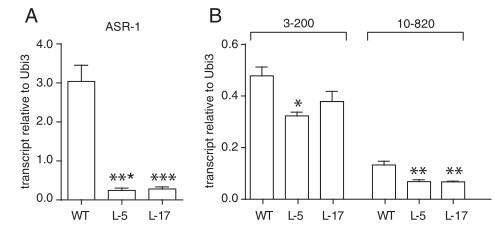
图四:靶基因的表达直接受ASR1调控
|








