|
细菌基因组denovo测序(框架图)技术介绍
***************************************************************************************************** 技术简介 细菌广泛存在于大自然中,其具有多样性丰富,基因组较小等特征。细菌是一类只存在拟核的裸露DNA的单细胞原核生物(基因组大小一般小于10Mb),它包括真细菌和古生菌两大类群,根据其基本形态可分为球菌,杆菌,螺旋菌,根据细胞壁组成成分,又分为革兰氏阳性菌和革兰氏阴性菌,大多数化脓性细菌属于革兰氏阳性菌,它们能产生外毒素使人致病;而大多数肠道菌则属于革兰氏阴性菌,它们能产生内毒素使人致病。常见的革兰氏阳性菌有葡萄球菌,链球菌,肺炎双球菌,破伤风杆菌等;常见的革兰氏阴性菌有痢疾杆菌,伤寒杆菌,大肠杆菌,变形杆菌等。 细菌对人类既有用处又有危害,一方面细菌是某些疾病的病原体,可导致伤寒、肺炎等疾病,另一方面,人类也常利用细菌制作乳酪及酸奶等。随着高通量测序技术的迅速发展,测序成本大幅降低使得获得具有简单基因组的细菌基因组信息更加便捷,获得某个细菌基因组信息,不但可以帮助了解其表型特征,致病机制以及其重要相关基因的功能,还可以通过和其它细菌基因组信息进行比对分析和进化分析,从而了解其分子进化机制。根据不同的研究目的和需求,细菌基因组测序又包括细菌de novo测序(扫描图、精细图、完成图)和细菌重测序。细菌基因组扫描图采用Illumina PE测序技术,对基因组进行~100×深度测序,根据序列组装结果,以获取基因组序列信息。 技术优势
b) 分析团队强大:拥有强大的生物信息团队,分析内容全面,并提供个性化分析 c) 实验体系严格:从DNA抽提,质检到文库构建,上机测序有严格的实验流程
b) 基因发现 c) 环境与生态研究 d) 疾病和个体化医疗
技术参数与实验流程
***************************************************************************************************** 技术参数
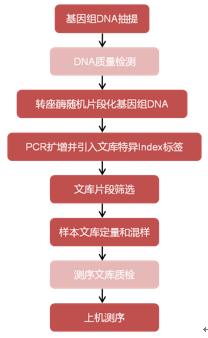
实验流程
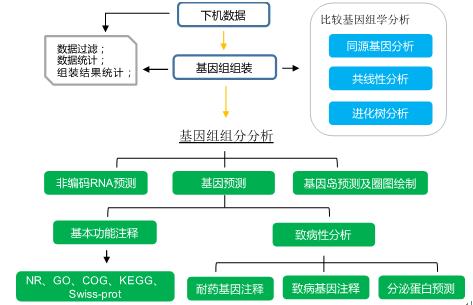
经典文章解读
*****************************************************************************************************案例:肠球菌Enterococcus faecium TX16的全基因组测序及比较分析 背景:在欧美国家,肠球菌是医院获得性感染(hospital-acquired infections)的主要因素,其中包括Enterococcus faecalis和 Enterococcus faecium两种菌株的感染。过去十年中,E. faecium菌株感染的比例不断上升。虽然在医院环境中E. faecium逐渐取代E. faecalis的机制并不清楚,但许多基因型鉴定和系统生物学研究已经指出了多种E. faecium菌株在全球临床环境中的广泛传播。然而,E. faecium完整基因组的缺失成为系统研究E. faecium的分子流行病学和发病机制的主要障碍。 方法:在本研究中,首次对E. faecium菌株TX16(隶属于ST18家族)进行了全基因组测序。随后将TX16的全基因组与21个E. faecium菌株基因组草图(未完全测通)进行了比对,通过系统生物学,多位点序列分析(MLST)和基因相似性分析,找到了医院相关菌株(HA)(hospital-associated strains,包含STs16、STs17、STs18、STs78家族)并对ORFs、多种基因和信号通路进行了分析。 结果: 1)HA菌株和非医院来源的CA菌株(community-associated)的核心基因组平均有3-4%的核苷酸序列差异; 2)378个ORFs在HA菌株中特异存在。大部份是转座子相关基因、载体基因、前噬菌体基因; 3)TX16基因组中找到9个与致病性相关的GIs; 4)抗生素抗药基因在HA菌株中大量富集; 5)可动因子(如IS16和转座子)也几乎是HA菌株独有的。 参考文献:Xiang Qin, et al. Complete genome sequence of Enterococcus faecium strain TX16 and comparative genomic analysis of Enterococcus faecium genomes. BMC Microbiology 2012, 12:135 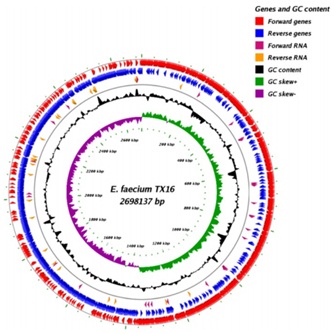
图一:肠球菌Enterococcus faecium TX16的全基因组圈图。TX16基因组含有2,698,137 bp,包含2,703个编码蛋白的ORFs、62个tRNA、6个拷贝的核糖体rRNA 和32非编码RNA。
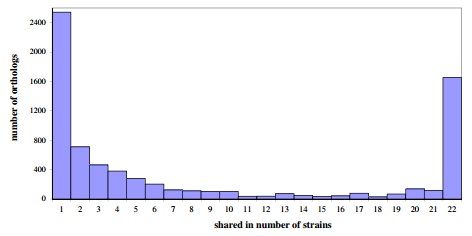
图二:22个E. faecium菌株的直接同源基因分析。分析发现2,543个基因只在某一个菌株中存在。22个菌株共有1,652个基因(1,608个单拷贝,44个多拷贝)。
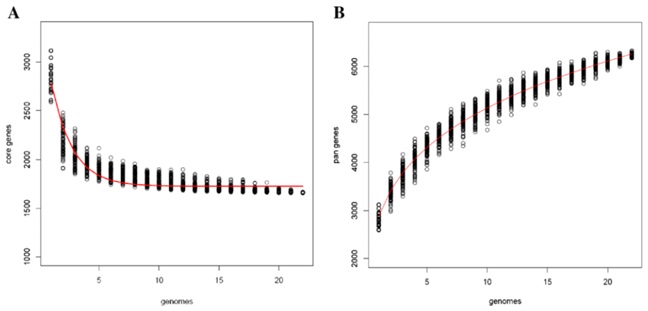
图三:22个E. faecium菌株的核心基因和全部基因数量。A、E. faecium核心基因数量,最后汇聚到1,726个基因;B、E. faecium全部基因数量,共包含6,262个基因。
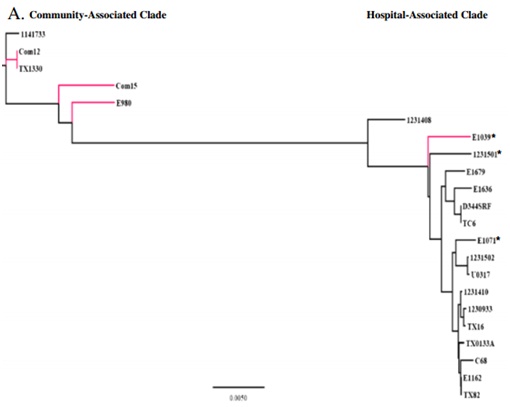
图四:22个E. faecium菌株的进化树。成功的将HA菌株与CA菌株区分开来。
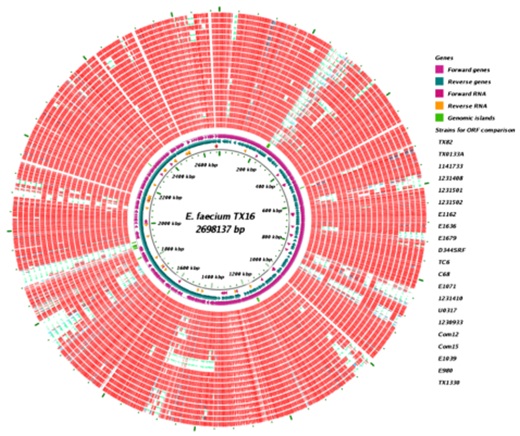
图五:22个E. faecium菌株基因组ORFs的比较。
|








