|
宏基因组测序技术介绍
***************************************************************************************************** 技术简介 宏基因组(metagenome)是指特定环境中全部生物(微生物)遗传物质DNA的总和。宏基因组测序研究就是通过在一个自然环境取样—一杯海水或一勺泥土,然后对样品内所有微生物DNA进行测序,其主要目的是分析环境样品所包含的全部微生物的群体基因组成及功能和参与的代谢通路,解读微生物群体的多样性与丰度,探求微生物与环境,微生物与宿主之间的关系,发掘和研究新的、具有特定功能的基因。 总的来说,宏基因组关注三个最核心的问题:微生物群落组成;挖掘基因的潜在功能活性;对基因进行功能注释。 应用领域
技术优势
技术参数与实验流程
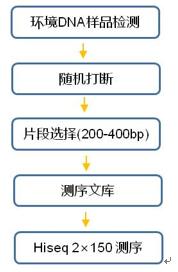
***************************************************************************************************** 技术参数
实验流程
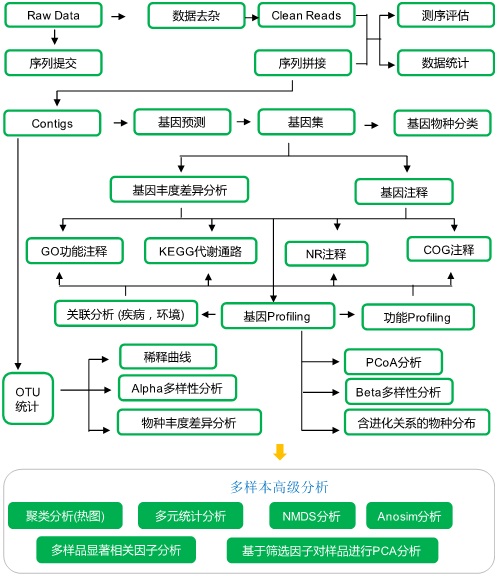
经典文章解读
***************************************************************************************************** 案例:断奶推动人类婴儿肠道微生物组成熟 背景:肠道微生物菌群和人类健康息息相关。但新生儿肠道微生物菌群的确立过程始终没有被系统的研究。本研究通过新生儿一年生长期的肠道微生物菌群宏基因组研究,探讨了新生儿肠道菌群变化,同时分析了生产方式和哺育方式对新生儿肠道菌群的影响。 方法:研究人员收集了瑞典98名母亲及其新生儿的粪便样本,然后在这些婴儿4个月和12个月的时候再次收集粪便样本,通过Illumina Hiseq平台宏基因组鸟枪测序技术测定肠道微生物的组成和功能基因。新生儿包括母乳喂养和非母乳喂养组别,顺产和破腹产组别。 结果:
参考文献:Backhed, F., et al., Dynamics and Stabilization of the Human Gut Microbiome during the First Year of Life. Cell Host Microbe, 2015. 17(5): p. 690-703. 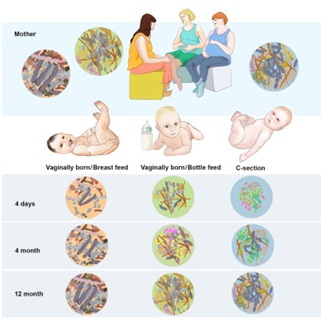
图一:本研究的摘要(图片版)
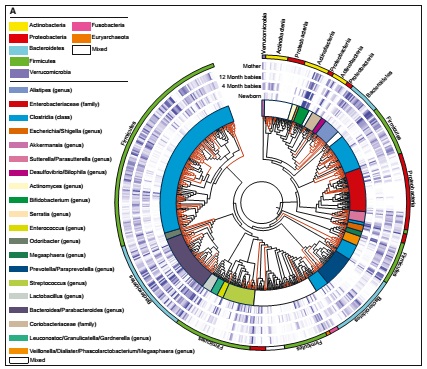
图二:基于625个META-OTU绘制母亲、新生儿不同生长期肠道微生物菌群进化树。
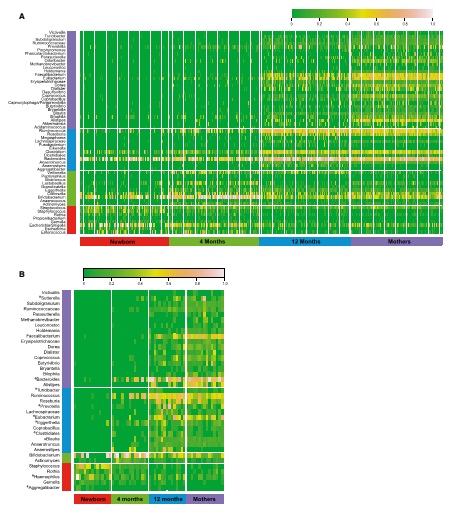
图三:不同样本来源的肠道微生物类群Heat-map。A:顺产婴儿与母亲;B:剖腹产婴儿和母亲。
|