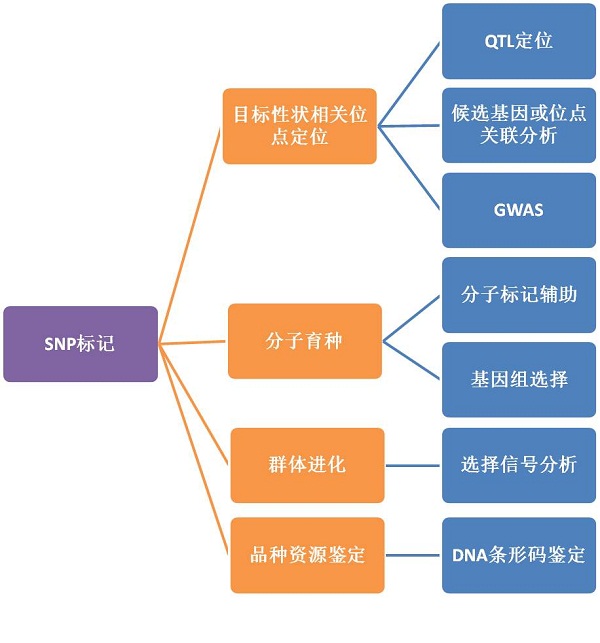
SNP是指在基因组上单个核苷酸的变异,SNP最重要的特点是数量很多,分布广泛,此外它还具有杂合率低、遗传稳定性好,便于高通量自动化监测分析等特点,因此SNP标记可作为目标性状相关位点定位(遗传图谱,QTL定位,GWAS)、分子育种 (基因组选择育种,分子标记辅助育种)、群体进化(选择信号分析)等研究的工具。
-
1. 目标性状相关位点定位---QTL定位
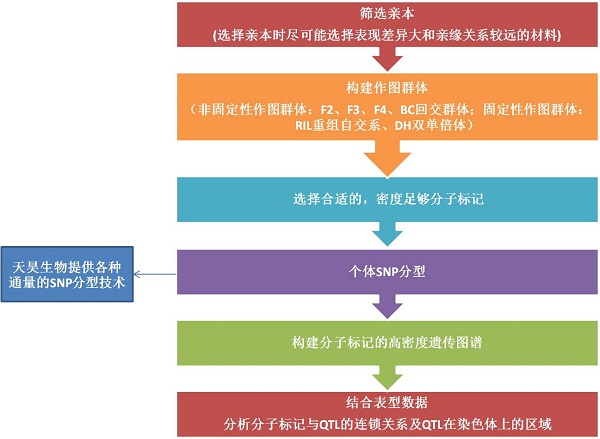
QTL,又称数量性状座位,一个QTL指在基因组中占据一定染色体区域,控制同一性状的一组微效多基因的集合或单个基因。但是目前的研究中大部分将QTL理解为单个基因(或主效基因)。 QTL的实质是通过分析整个染色体组的遗传标记和数量性状表型值的关系,将一个或多个QTL定位到位于同一染色体的遗传标记旁。目前,基于全基因组重测序的混池测序方法可以对质量性状单基因和数量性状主效基因进行快速定位。
QTL定位的传统研究路线
基于高通量测序的QTL定位研究路线
-
2. 目标性状相关位点定位---候选基因或位点关联分析
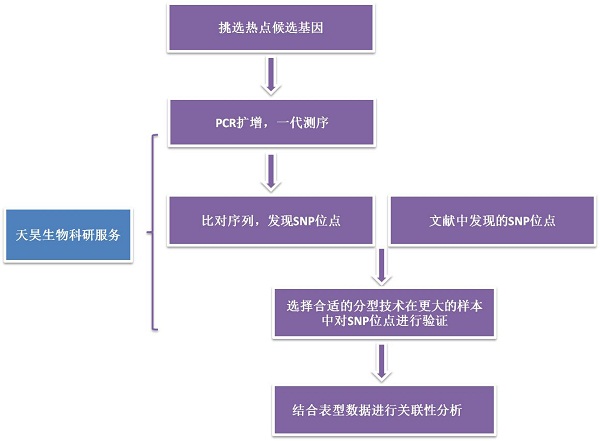
关联分析,亦称作连锁不平衡作图,这种方法主要基于两个位点间等位基因的非随机共分离现象,利用群体内的原始重组和自然变异来研究数量性状。与基于两个亲本杂交形成的作图群体的QTL定位相比,关联分析侧重于研究个体之间彼此并无关系的自然群体,因此比连锁分析具有更高的作图精度,可以直接找到目标基因变异,因而被广泛应用于复杂数量性状的遗传分析。关联分析可以分为全基因组关联分析和候选基因或位点关联分析,其中候选基因或位点关联分析是对性状相关调节通路关键酶基因或位点进行分子标记的关联分析研究。通过关联分析发掘的功能型分子标记可应用于分子遗传育种中的分子标记辅助选择(MAS)。
候选基因或位点关联分析的研究路线
-
3. 目标性状相关位点定位---全基因组关联分析
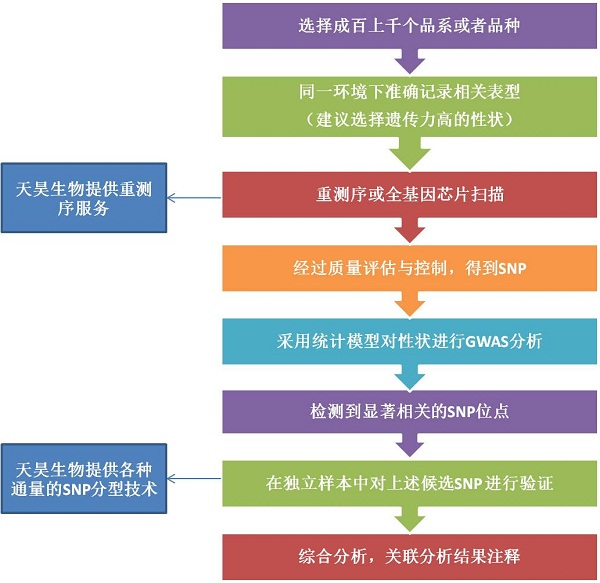
全基因组关联分析(GWAS)是对遗传多样性丰富的自然群体的每个个体进行全基因组重测序或者全基因芯片扫描,结合表型数据采用数据模型进行全基因组关联分析,从而获得目标性状关联位点或者候选基因。目前GWAS已经成为动植物多个复杂性状同时进行精细定位的有力工具,在全基因组范围内筛选有可能会与农艺性状(株高、分蘖数、穗粒数、千粒重等)和经济性状(肉质、产奶量、产蛋量、产肉量等)相关的变异位点,能够为下一步基因的功能研究、标记辅助选择等工作提供分子理论依,同时为进一步培育优质的品系积累分子资料。
GWAS定位的研究路线
-
4. 分子育种—分子标记辅助
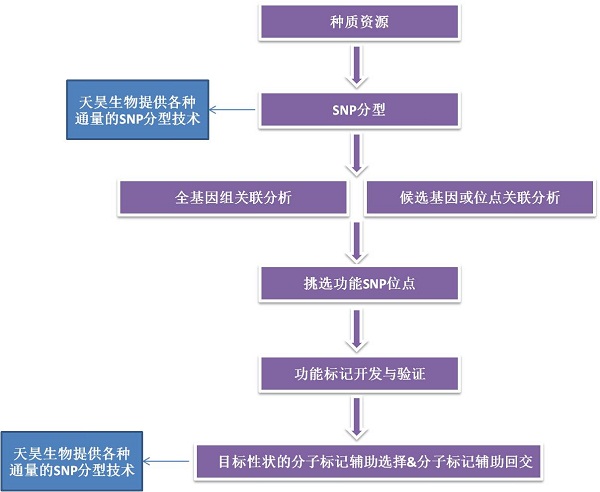
传统育种首先是通过各种途径创造遗传变异,然后从分离群体的后代中进行优化选择和评价。因此传统育种选择是建立在植株的表型基础上,这就要求育种工作者必须有丰富的实践经验。分子标记辅助选择(MAS) 不直接利用性状本身信息,而是利用分子标记与决定目标性状基因紧密连锁的特点,通过检测分子标记,即可检测到目的基因的存在,达到选择目标性状的目的,具有快速、准确、不受环境影响的优点,从而加快育种速度,节省田间试验开支,对于低遗传力性状和难以测量的性状具有更大的优势。可应用于目标性状的分子标记辅助选择和分子标记辅助回交。
-
1. 目标性状的分子标记辅助选择
利用与目标性状紧密连锁的SNP标记对育种后代的分离群体进行选择,例如一个分子标记的等位基因X与小麦白叶枯病抗病性紧密连锁,等位基因Y与感病性连锁,如果育种后代个体检测到等位基因X,则这个个体可能对小麦白叶枯病产生抗病性。
-
2. 分子标记辅助回交
标记辅助回交又称标记辅助导入,即将与目标性状相关联的基因从供体材料导入到受体育种材料最有效的技术路径,适用于自交系或品种的性状改良; 不但可以对导入片段进行选择,又可加速轮回亲本基因组的恢复即背景选择,即依靠标记对后代个体进行全基因组选择,加速育种进程,提高选择效率。
分子标记辅助的研究路线
-
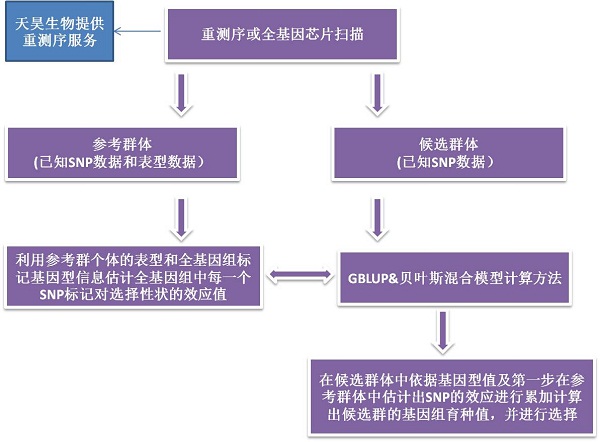
5. 分子育种—基因组选择
分子标记辅助只关注主效基因带来的变异,而小效应累加起来所带来的变异却被忽视了,为了捕获构成表型的所有遗传变异,需要在基因组水平上检测影响目标性状的所有QTL 并对其加以利用,这就是全基因组选择(GS)。基因组选择是利用具有表型和基因型的个体来预测只具有基因型不具有表型值动物的基因组育种值(GEBV),育种值高,该个体所含的优良基因可遗传的物质就多,产生优良后代的可能性越高。基因组选择优势是它能够在得到动物刚出生时即对其进行育种值评估,因此利用基因组选择可以缩短世代间隔,从而加快遗传进展并且降低经济投入。
全基因组选择主要利用的是连锁不平衡信息,即假设每个标记与其相邻的QTL 处于连锁不平衡状态,因而利用标记估计的染色体片段效应在不同世代中是相同的。由此可见,标记的密度必须足够高,以确保控制目标性状的所有的QTL 与标记处于连锁不平衡状态。 另外遗传力高低,表型记录个体数 ,参考群体与候选群体间的关系 以及参考群体内的亲缘关系都会影响基因组选择的准确度。
基因组选择的研究路线
-
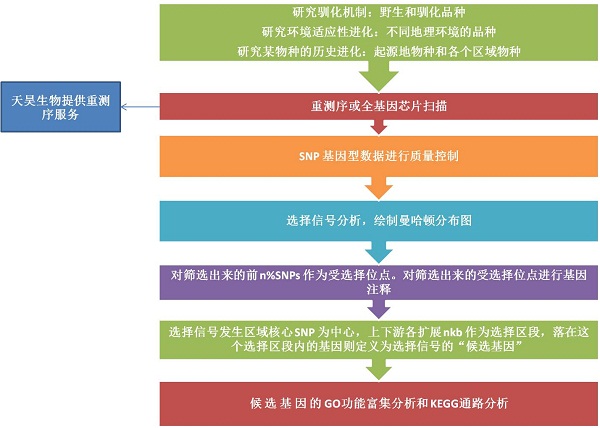
6. 群体进化—选择信号分析
选择即是在世代的传递过程中,因为自然选择,人工驯化等因素会使某种基因型个体的比例发生变化的群体遗传学现象。在选择的作用下,群体有利等位基因频率则会在短时间内达到一个较高的值,同时选择作用下的连锁不平衡会造成选择位点附近的中性位点的基因频率随之增加形成长范围的单倍型纯合,群体遗传学中,将这种由选择作用造成的部分染色体片段的多态性降低称为选择性扫除,与选择相对应的基因组信息被称为“选择信号”,选择性扫除是选择在基因组上留下的明显特征,一般表现为基因的纯合以及某些位点或 DNA 片段多态的降低。研究人工选择不仅能够探究影响品种多样性的潜在遗传机制,而且还能精细定位部分受到选择且与质量性状相关的功能基因或者与重要经济性状相关的主效基因,这是因为选择信号潜在区域多与性状相关的重要候选基因重叠,因此基因组选择信号的检测对研究物种驯化过程和开展育种规划具有非常重要的现实意义。
选择信号分析的研究路线
-
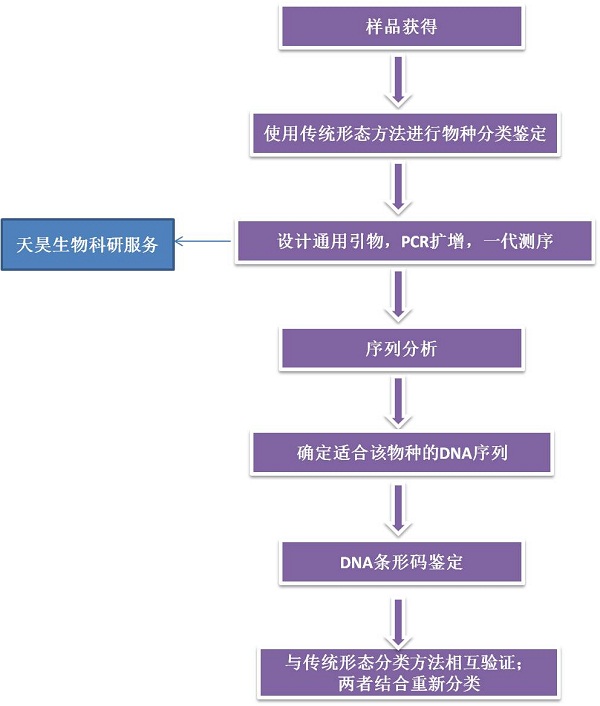
7. 品种资源鉴定—DNA条形码鉴定
DNA 条形码技术(DNA barcoding)是利用有足够变异的、 易扩增且相对较短的 DNA 片段自身在物种种内的特异性和种间的多样性而创建的一种新的生物身份识别系统,它可以不受形态特征和生物个体发育时期的限制,能够对物种进行快速的鉴定,DNA 条形码在物种鉴定与分类、种群遗传、快速查验有害生物及入侵种、发现新物种和保护生物多样性、濒危物种保护等研究领域具有广泛的应用前景。在动物中COI基因常被用作DNA条形码,在植物中3个质体基因 (rbcL, matK和 trnH-psbA) 以及核糖体基因 (ITS) 常被用作DNA条形码,但是一般会根据所研究的物种选择最合适的DNA条形码组合,因为这些常用的DNA条形码也有PCR扩增率低,引物通用性差等问题。
DNA条形码鉴定的研究路线
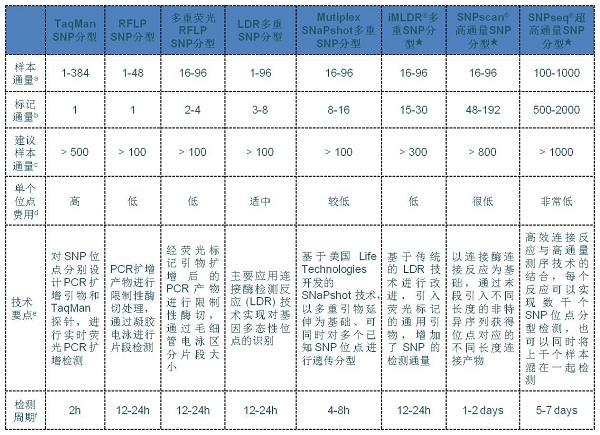
天昊生物提供多种创新的SNP检测方法,涵盖了从低通量、中等通量到高通量的完整SNP分型平台,客户可根据实验项目的样本和分子标记规模选择最优的SNP检测技术。
a:单次实验可同时检测的样本 b:单次实验可同时检测的分子标记数目 c:控制最优实验成本所需的最小样本起始量 d:单个样品单次检测中一个位点的实验成本 e:检测方法的技术基础和平台 f:单次实验从DNA到实验结果解读的时间 ★ 天昊生物的创新技术
|